SVD, pseudoinverse en PCA: SVD, pseudoinverse en PCA in Python
 SVD en beeldcompressie
SVD en beeldcompressie
R heeft de functie svd aan boord in het linalg pakket om numeriek de singulierewaardenontbinding te bepalen.
Eerst rekenen we het voorbeeld uit de lestekst na:
>>> import numpy as np
>>> import numpy.linalg as la
>>> A = np.array([[2,0],[0,-3]]); print(A)
[[ 2 0]
[ 0 -3]]
>>> U, S, Vt = la.svd(A, full_matrices=True) # singulierewaardenontbinding (SVD) van A
>>> print(U) # matrix U in SVD
[[0. 1.]
[1. 0.]]
>>> print(np.diag(S)) # diagonaalmatrix met singuliere waarden van A
[[3. 0.]
[0. 2.]]
>>> print(Vt) # getransponeerde van matrix V in SVD
[[-0. -1.]
[ 1. 0.]]
>>> print(U @ np.diag(S) @ Vt - A) # controle van svd(A)
[[0. 0.]
[0. 0.]]
Het volgende voorbeeld van een singuliere matrix illustreert het verschil tussen exact en numeriek rekenen. \[\begin{aligned}\matrix{1 & -1\\ -1 & 1} &= \matrix{-\frac{1}{2}\sqrt{2} & \frac{1}{2}\sqrt{2} \\ \frac{1}{2}\sqrt{2} & \frac{1}{2}\sqrt{2}}\matrix{2 & 0\\ 0 & 0}\matrix{-\frac{1}{2}\sqrt{2} & \frac{1}{2}\sqrt{2} \\ -\frac{1}{2}\sqrt{2} & -\frac{1}{2}\sqrt{2}}\\ \\ &=\frac{1}{2}\sqrt{2} \matrix{-1 & 1\\ 1 & 1}\matrix{2 & 0\\ 0 & 0}\matrix{-1 & 1\\ -1 & -1}\frac{1}{2}\sqrt{2} \end{aligned} \]
>>> B = np.array([[1, -1],[-1,1]]); print(B)
[[ 1 -1]
[-1 1]]
>>> U, S, Vt = la.svd(B, full_matrices=True) # singulierewaardendenontbinding
>>> print(U) # matrix U in SVD
[[-0.70710678 0.70710678]
[ 0.70710678 0.70710678]]
>>> print(np.diag(S)) # matrix met singuliere waarden op hoofddiagonaal
[[2.00000000e+00 0.00000000e+00]
[0.00000000e+00 3.35470445e-17]]
>>> print(Vt) # getransponeerde van matrix V in SVD
[[-0.70710678 0.70710678]
[-0.70710678 -0.70710678]]
>>> print(U @ np.diag(S) @ Vt - B) # controle van svd(B)
[[-3.33066907e-16 4.44089210e-16]
[ 0.00000000e+00 -2.22044605e-16]]
>>> print(np.round(U @ np.diag(S) @ Vt - B, 3)) # afgerond
[[-0. 0.]
[ 0. -0.]]
Nu een voorbeeld van beeldcompressie aan de hand van een patroon bestaande uit witte en zwarte hokjes op een \(9\times 9\) rooster. We specificeren de figuur met een matrix bestaande uit nullen (voor witte hokjes) en enen (voor zwarte hokjes): \[M = \matrix{ 0 & 0 & 0 & 0 & 1 & 0 & 0 & 0 & 0\\ 0 & 0 & 0 & 1 & 0 & 1 & 0 & 0 & 0 \\ 0 & 0 & 1 & 0 & 0 & 0 & 1 & 0 & 0\\ 0 & 1 & 0 & 1 & 1 &1 & 0 & 1 & 0 \\ 1 & 0 & 0 & 1 & 0 & 1 & 0 & 0 & 1\\ 0 & 1 & 0 & 1 & 1 &1 & 0 & 1 & 0 \\ 0 & 0 & 1 & 0 & 0 & 0 & 1 & 0 & 0\\ 0 & 0 & 0 & 1 & 0 & 1 & 0 & 0 & 0 \\ 0 & 0 & 0 & 0 & 1 & 0 & 0 & 0 & 0}\] In R kunnen we dit patroon gemakkelijk visualiseren.
>>> import numpy as np
>>> import numpy.linalg as la
>>> import matplotlib.pyplot as plt
>>> M = np.array(
[[0, 0, 0, 0, 1, 0, 0, 0, 0],
[0, 0, 0, 1, 0, 1, 0, 0, 0],
[0, 0, 1, 0, 0, 0, 1, 0, 0],
[0, 1, 0, 1, 1, 1, 0, 1, 0],
[1, 0, 0, 1, 0, 1, 0, 0, 1],
[0, 1, 0, 1, 1, 1, 0, 1, 0],
[0, 0, 1, 0, 0, 0, 1, 0, 0],
[0, 0, 0, 1, 0, 1, 0, 0, 0],
[0, 0, 0, 0, 1, 0, 0, 0, 0]]) # patroon
>>> plt.imshow(np.ones_like(M) - M, cmap='gray')
>>> plt.show()
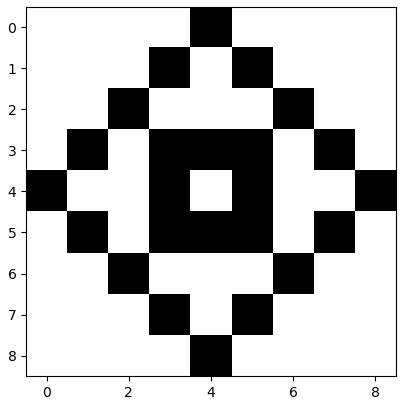
Stel nu dat we iemand dit patroon willen sturen. Dan kunnen we de matrix \(M\) sturen, oftewel 81 getallen nul en één. Maar met de singulierewaardenontbinding kun je minder getallen sturen, terwijl het plaatje toch gereconstrueerd kan worden.
De eigenwaarden van de matrix \(M\) zijn in dalende volgorde als volgt:
>>> print(np.flip(np.round(np.sort(vals), 3)))
[ 3.709 2. 1.194 0. 0. -0. -0. -0.903 -2. ]
Dus zijn de singuliere waarden van \(M\) in opklimmende volgorde, afgerond op 1 decimaal, gelijk aan \(3.7\), \(2.0\), \(1.2\), \(0.9\) en \(0\).
We benaderen \(M\) nu met de hoogste termen van de singulierewaardenontbinding. Maar in plaats van de numerieke waarden van de ontbinding te gebruiken voor beeldweergave ronden we matrixelementen af op gehele getallen \(0\) en \(1\) om het patroon te reconstrueren.
Stel dat we alleen de hoogste singuliere waarde \(3.7\) gebruiken. Dan lijkt het plaatje al wat op de oorspronkelijke figuur:
>>> U, S, Vt = la.svd(M, full_matrices=True) # singulierewaardenontbinding (SVD) van M
>>> S1 = np.zeros((9,9)); S1[0,0] = S[0]
>>> M1 = np.round( U @ S1 @ Vt)
>>> plt.imshow(np.ones_like(M1) - M1, cmap='gray')
>>> plt.show()
Hiervoor hoeven we dus maar \(1+2\times 9 = 19\) numerieke waarden op te sturen, namelijk de singuliere waarde en de twee vectoren \(\vec{u}_1\) en \(\vec{v}_1\) .
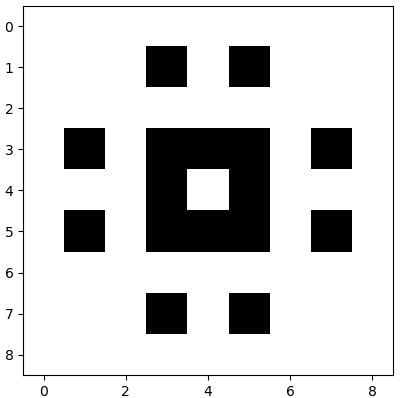
Als we ook de singuliere waarde \(2\) gebruiken, dan krijgen we de oorspronkelijke figuur terug.
>>> S3 = np.zeros((9,9)); S3[0,0] = S[0]; S3[1,1] = S[1]; S3[2,2] = S[2]
>>> M3 = np.round(U @ S3 @ Vt)
>>> plt.imshow(np.ones_like(M3) - M3, cmap='gray')
>>> plt.show()
Nu volstaat het opsturen van \(3+6\times 9 = 57\) numerieke waarden, namelijk \(3\) eigenwaarden en \(2\times 3= 6\) vectoren. Dit lijkt niet zo veel minder dan \(81\), het gaat toch al om een reductie van ongeveer \(30\%\)
\(\phantom{xx}\)
De winst is spectaculairder bij het sturen van grotere digitale beelden, bijvoorbeeld op de in computer vision veel gebruikte foto van \(512\times 512\) pixels. Hieronder tekenen we de assen erbij.
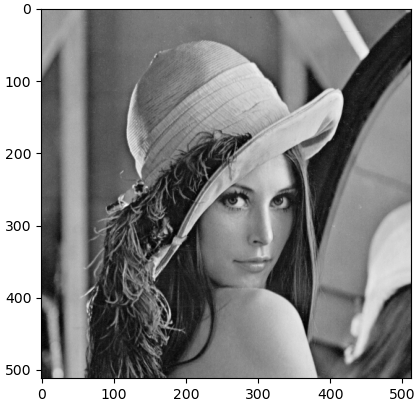
We lezen de foto in, berekenen de singulierewaardenontbinding van de bijpassende (\(512\times 512\))-matrix met grijswaarden, en benaderen de originele foto met 8, 16, 32, 64, en 128 termen uit de ontbinding.
>>> import numpy as np
>>> import numpy.linalg as la
>>> import matplotlib.pyplot as plt
>>> from imageio import imread
>>> img = imread('D:/lena512.bmp') # laad de foto
>>> plt.imshow(img, cmap='gray')
>>> plt.show() # toon de foto met assen
>>> [U,S,Vt] = la.svd(img) # singulierewaardenontbinding
>>> img8 = U[:,0:8] @ np.diag(S[0:8]) @ Vt[0:8,:] # beeldcomppressie k= 8
>>> img16 = U[:,0:16] @ np.diag(S[0:16]) @ Vt[0:16,:] # beeldcomppressie k= 16
>>> img32 = U[:,0:32] @ np.diag(S[0:32]) @ Vt[0:32,:] # beeldcomppressie k= 32
>>> img64 = U[:,0:64] @ np.diag(S[0:64]) @ Vt[0:64,:] # beeldcomppressie k= 64
>>> img128 = U[:,0:128] @ np.diag(S[0:128]) @ Vt[0:128,:] # beeldcomppressie k=128
>>> fig = plt.figure() # toon foto's in een 3x2 rooster
>>> fig.add_subplot(3,2,1)
>>> plt.axis('off')
>>> plt.imshow(img8, cmap='gray')
>>> fig.add_subplot(3,2,2)
>>> plt.axis('off')
>>> plt.imshow(img16, cmap='gray')
>>> fig.add_subplot(3,2,3)
>>> plt.axis('off')
>>> plt.imshow(img32, cmap='gray')
>>> fig.add_subplot(3,2,4)
>>> plt.axis('off')
>>> plt.imshow(img64, cmap='gray')
>>> fig.add_subplot(3,2,5)
>>> plt.axis('off')
>>> plt.imshow(img128, cmap='gray')
>>> fig.add_subplot(3,2,6)
>>> plt.axis('off')
>>> plt.imshow(img, cmap='gray')
>>> plt.show()
Hieronder zie je de beelden voor verschillende getoonde waarden van het aantal termen \(k\) die uit de singulierewaardenontbinding gehaald worden. Duidelijk is dat je minder informatie nodig hebt om toch een foto te hebben die op het zicht weinig afwijkt van het origineel (bij \(k=64\) is de compressiefactor ongeveer \(\frac{1}{4}\)). Bij kleurenfoto's is de winst zelfs groter.
| \(k=8\) | \(k=16\) |
 |
 |
| \(k=32\) | \(k=64\) |
 |
 |
| \(k=128\) | origineel: \(k=512\) |
 |
 |


